그림이나 수식이 깨질 경우 링크에서 확인하세요.
Introduction
병리 슬라이드는 cell의 morphology를 보여줄 뿐 아니라 면역조직화학염색(immunohistochemistry staining)과 같은 방법을 통해 유전자의 발현 상태까지 보고 진단을 내리고 치료 방침을 결정할 수 있습니다. 기본적으로 여지껏 진행되어왔던 인공지능 병리학 연구는 cell morphology에 기반한 방법론들이 대부분인데, 이는 gene expression과 같은 또다른 정보를 잘 encoding할 수 없었기 때문입니다.
또한 병리 이미지의 사이즈는 GPU의 VRAM에 올리기에는 너무나도 거대하기 때문에, 주로 과거의 연구들은 병리 이미지를 patch로 잘라서 patch 단위의 labeling을 하곤 했죠. 하지만 이 역시 지나치게 labor-intensive하며 현실적으로 large, bulk dataset을 만들기가 불가능에 가깝습니다.
이러한 문제들을 해결하고자 본 논문에서는 self-supervised learning(SSL)을 이용한 multiple instance learning(MIL)을 통해 patch단위의 embedding 정보 뿐 아니라 저자들이 slice embedding이라고 말하는 embedding까지 추출할 수 있었으며, 이를 gene expression 정보와 align 할 수 있기까지 했다고 합니다.
Self-supervised learning (SSL)
SSL은 representation learning의 한 종류로, 이미지의 feature를 잘 배울 수 있게 하는 방법론입니다. 이를 도식화하면 다음과 같은데 이는 variational autoencoder의 모식도와 비슷하지만, 방향만 반대인 것으로 이해할 수 있습니다.
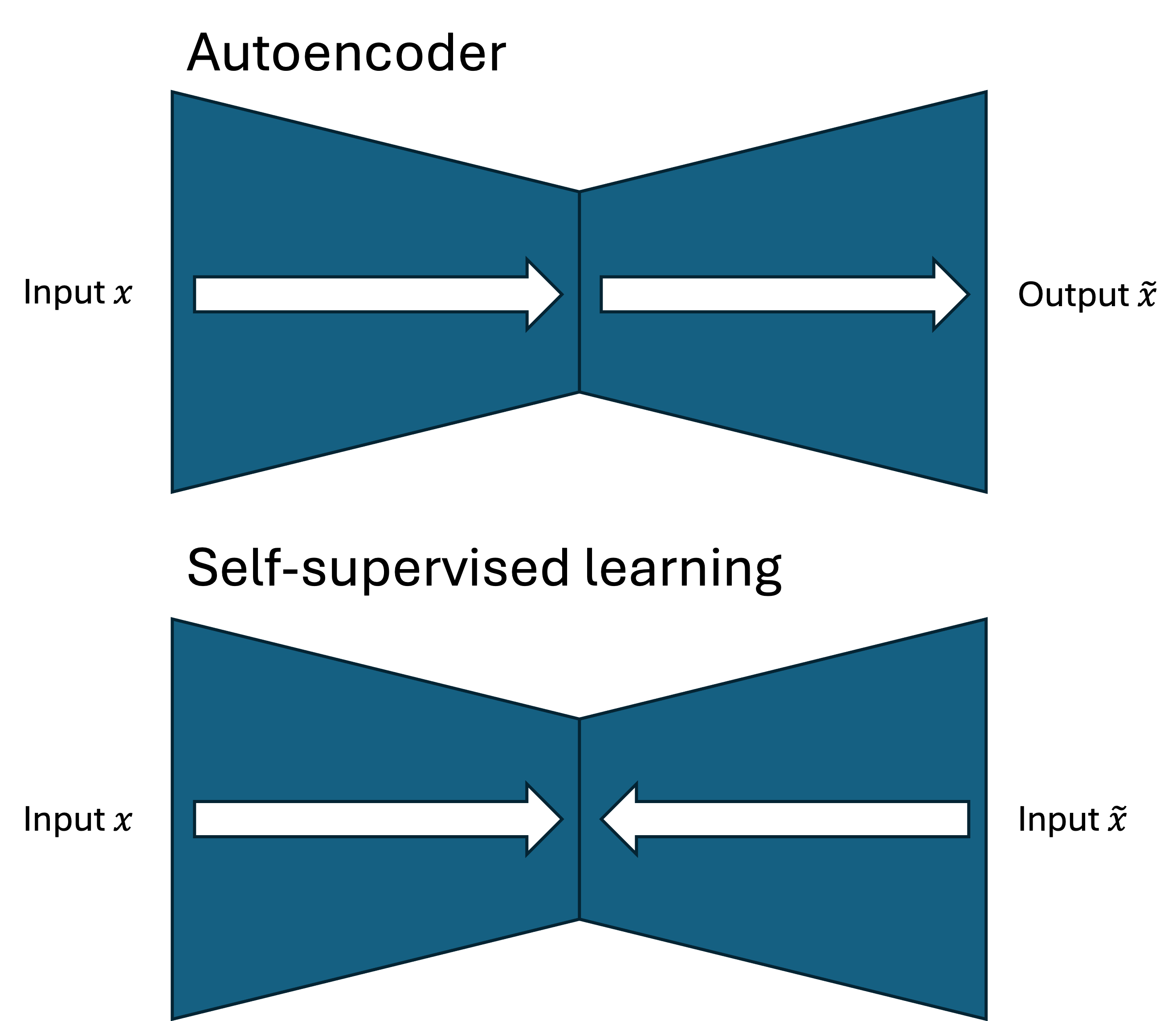
Autoencoder에서는 input $x$를 encoder과 decoder에 넣어서 output $\widetilde{x}$를 만들어내고, 이 둘을 비슷하게 만들어내는 과정을 수행한다면,
SSL에서는 input $x$와 이를 살짝 변형시킨(augmentation한) $\widetilde{x}$를 (주로) 동일한 encoder에 넣어서 latent feature $z$와 $\widetilde{z}$로 만든 다음, 이 latent variable들의 similarity를 극대화하는 방법론을 사용합니다.
이에 대한 수학적, 인과적 formulation은 DeepMind에서 내놓은 RELIC 논문에서 확인할 수 있는데, 모식도는 다음과 같습니다.

우리가 이미지를 인식할 때, 우리 뇌는 본능적으로 image($X$)의 content($C$)와 style($S$)를 구분하여 인식합니다. 예를 들어, 흑백 이미지와 컬러 이미지의 사람 얼굴이 있다면 우리는 사람 얼굴($C$)는 동일하게 인식하며 흑백과 컬러라는 이미지의 스타일($S$)은 무시하고 인식할 수 있습니다.
따라서 딥러닝 모델이 이미지의 표현(representation)을 잘 인식한다는 말은 $S$는 최대한 무시하고, $C$만을 추출하여 downstream task인 $Y_i$들을 잘 처리해낼 수 있게 된다는 말이지요. 이것이 SSL의 이론적 배경이 되는 것입니다. 이를 수식화하면 다음과 같습니다.
\[p^{do(S=s_i)}(Y|C)=p^{do(S=s_j)}(Y|C)\]Style이 $s_i$냐 $s_j$냐에 상관없이 content $C$가 들어왔을 때 output $Y$가 나올 확률은 동일해야 한다는 식입니다.
이렇게 SSL을 간단하게 이해해볼 수 있습니다.
Methods
Slide encoder
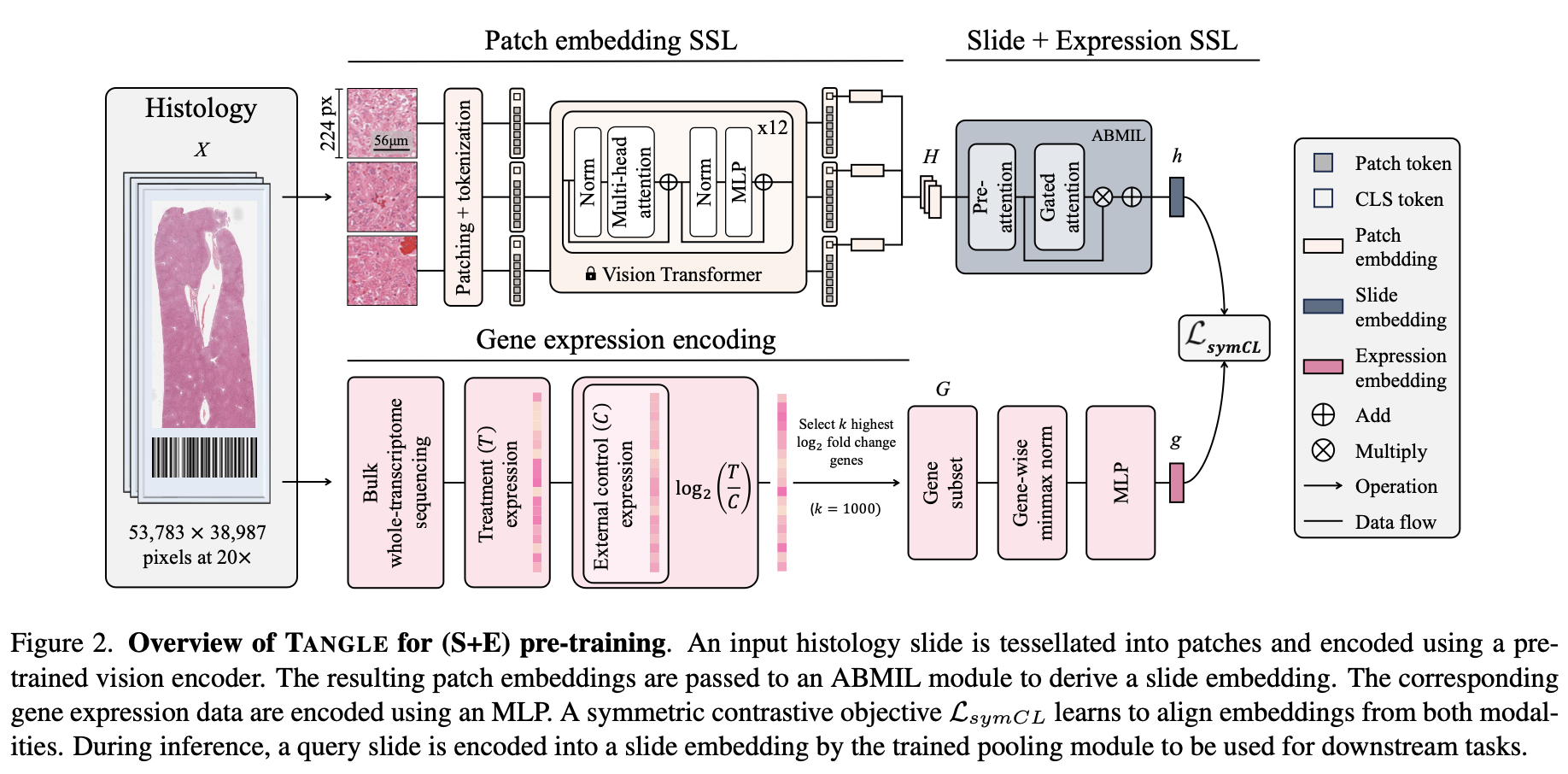
RGB histology slide $X_i\in\mathbb{R}^{d_x\times d_y\times 3}$이 있을 때 저자들은 MIL methodology를 사용하여 모델을 학습시켰습니다.
Pre-trained patch encoding: 인간 조직을 encoding 하기 위해서, 저자들은 CTransPath라는 모델을 사용하였습니다. 이 pretrained model에 대해서 slide $X_i$가 encoding된 output을 $H_i\in\mathbb{R}^{N_\mathcal{H}\times d_{\mathcal{H}}}$라고 합시다. 이 때 $N_{\mathcal{H}}$는 patch embedding의 개수이고 $d_{\mathcal{H}}$는 그 차원입니다.
MIL slide encoding: Patch embedding $H_i\in\mathbb{R}^{N_\mathcal{H}\times d_{\mathcal{H}}}$을 slide embedding $h_i\in\mathbb{R}^{d}$로 만들기 위해서 저자들은 Attention-based MIL model (ABMIL) 을 사용하였습니다.
Gene expression encoder
Raw transcroptomic measurement들이 $N_{\mathcal{G}}$개의 유전자에 대해 있을 때, 저자들은 $\log 2$ fold change를 대조군에 대해서 계산하였습니다. 이 slide $X_i$에 대한 $\log 2$ fold change를 $t_i\in\mathbb{R}^{N_{\mathcal{G}}}$로 씁니다. 이는 multilayer perceptron (MLP) $\phi:\mathbb{R}^{\mathcal{N_{\mathcal{G}}}}\to\mathbb{R}^d$를 통해 encoding이 가능합니다. 이제 $\phi(t_i)=g_i$로 씁시다.
Multimodal alignment
Pre-training contrastive alignment: 위와 같이 얻은 두 가지 embedding vector, slide embedding과 expression encoder를 symmetric cross-modal contrastive learning objective를 통해 맞춥니다. 이는 다음처럼 정의됩니다:
\[\mathcal{L}_{SymCL}=-\frac{1}{2M}\sum_{j=1}^M\log\frac{\exp(\tau h_i^Tg_i)}{\sum_{i=1}^M\exp(\tau h_i^Tg_j)}\\-\frac{1}{2M}\sum_{j=1}^M\log\frac{\exp(\tau g_j^Th_j)}{\sum_{j=1}^M\exp(\tau g_j^Th_i)}\]이 때 첫 번째 항은 slide-to-expression 항이고, 두 번째 항은 expression-to-slide 항입니다. 각 항은 embedding 사이의 similarity를 최적화합니다.
Complementary objective: contrastive loss의 대안으로, expression construction loss $\mathcal{L}{REC}$과 vision-only intra-modaality loss $\mathcal{L}{INTRA}$도 제안합니다. 전자는 expression regression task를 표현해주는 loss이고, 후자는 slide의 random subset (local-to-local) loss를 align 해줍니다. 이들은:
\[\mathcal{L}_{REC}=\frac{1}{M}\sum_{i=1}^M\|g_i-c(f(H_i))\|_2\]와
\[\mathcal{L}_{INTRA}=-\frac{1}{2M}\sum_{i=1}^M\log(\text{Softmax}(h_{i,1}^T\overline{h}_i,\tau))\\ -\frac{1}{2M}\sum_{i=1}^M\log(\text{Softmax}(h_{i,1}^Th_{i,2},\tau))\]로 정의됩니다. 이 때 $c(\cdot)$은 MLP regressor이고, $\overline{h}i$는 average patch embedding $\overline{h}_i=\frac{1}{N{\mathcal{H}}^{(i)}}\sum_jH_{ij}^{N_{\mathcal{H}}^{(i)}}$이고, $h_{i,1}$과 $h_{i,2}$는 다른 random patch embedding subset에서 추출된 slide embedding입니다. 이들을 TANGLE-REC과 INTRA로 각각 부를 것입니다.
이 과정을 도식화하면 다음과 같습니다.
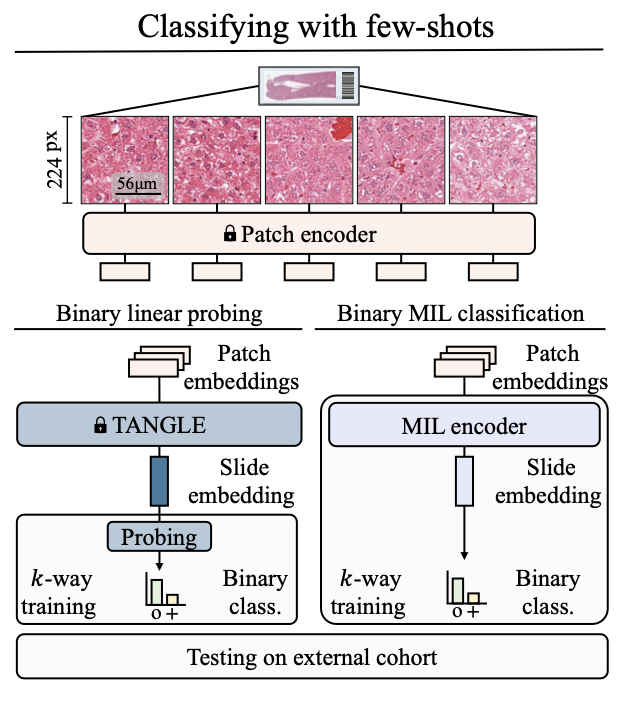
Inference: inference시에는 query slide가 vision encoder를 통과해서 patch embedding을 뽑아내고, MIL module에 들어가서 molecular signiture에 해당하는 morphological manifestation에 대한 slide embedding을 뽑아냅니다.
이 과정을 도식화하면 다음과 같습니다.
Experiments
저자들은 이렇게 학습한 TANGLE을 downstream task에 대해서 성능 확인을 하였습니다.
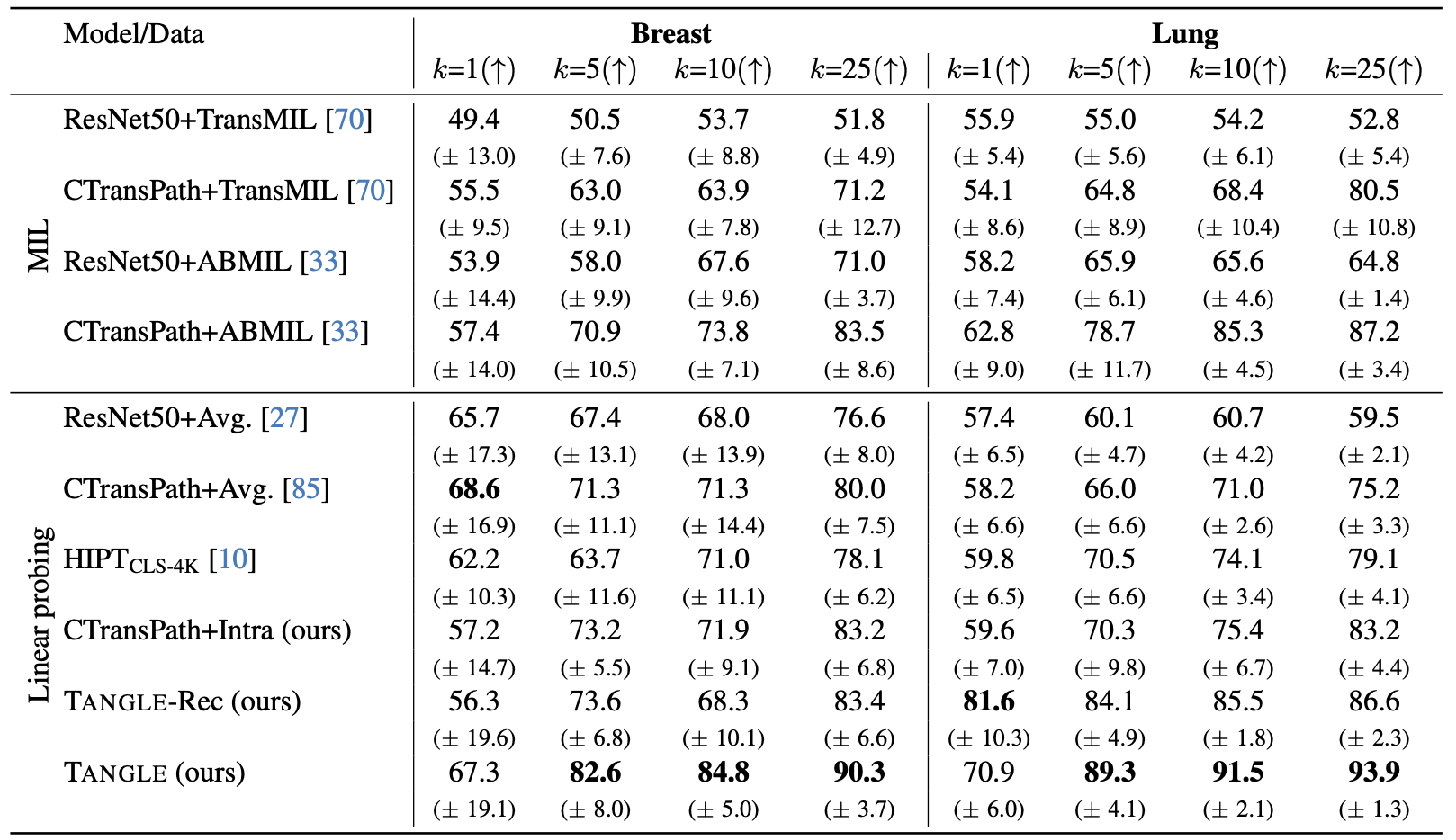
Human breast and lung에 대한 few-shot cancer subtype classification의 AUC score들입니다. Breast slide 1,265장과 lung slide 1,946장에 대한 성능을 기록하였습니다. TANGLE이 더 좋은 성능을 냄을 확인할 수 있습니다.
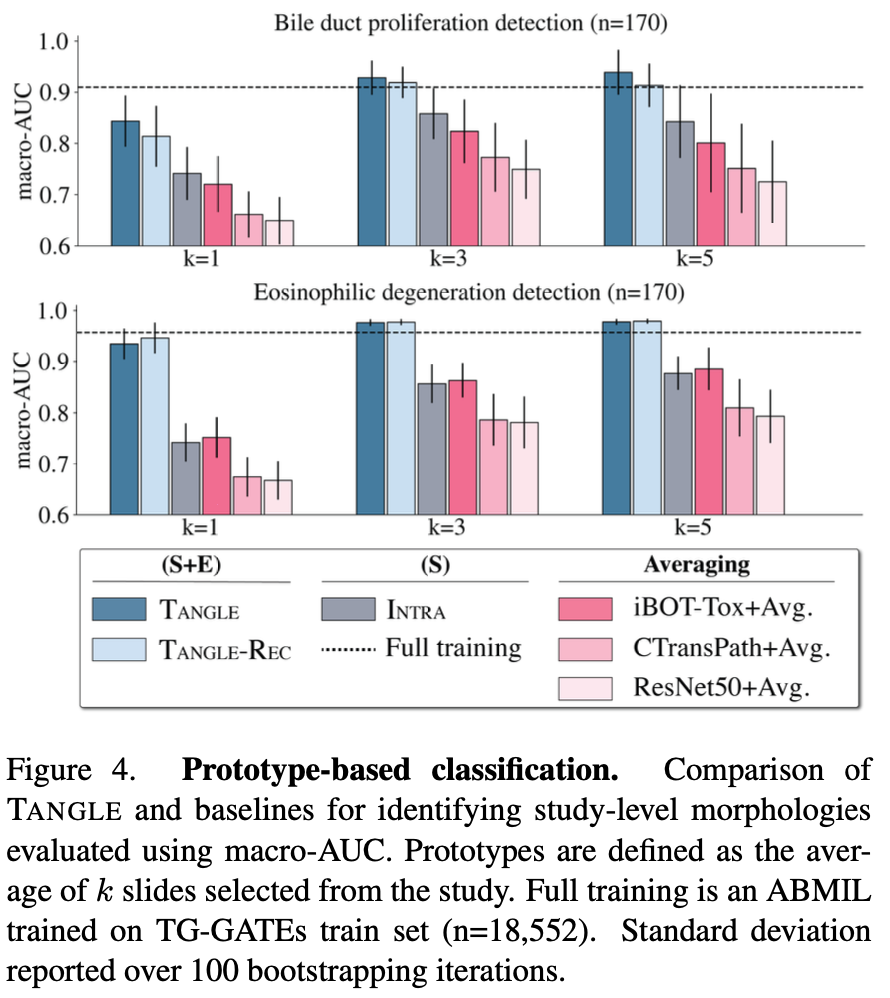
Study-level morphology에 대한 macro-AUC 성능입니다. TANGLE이 full traning인 ABMIL과 유사한 성능을 보임을 확인할 수 있습니다.

Slide retrieval에 대한 성능입니다. TANGLE이 가장 높은 성능을 보임을 확인할 수 있습니다.
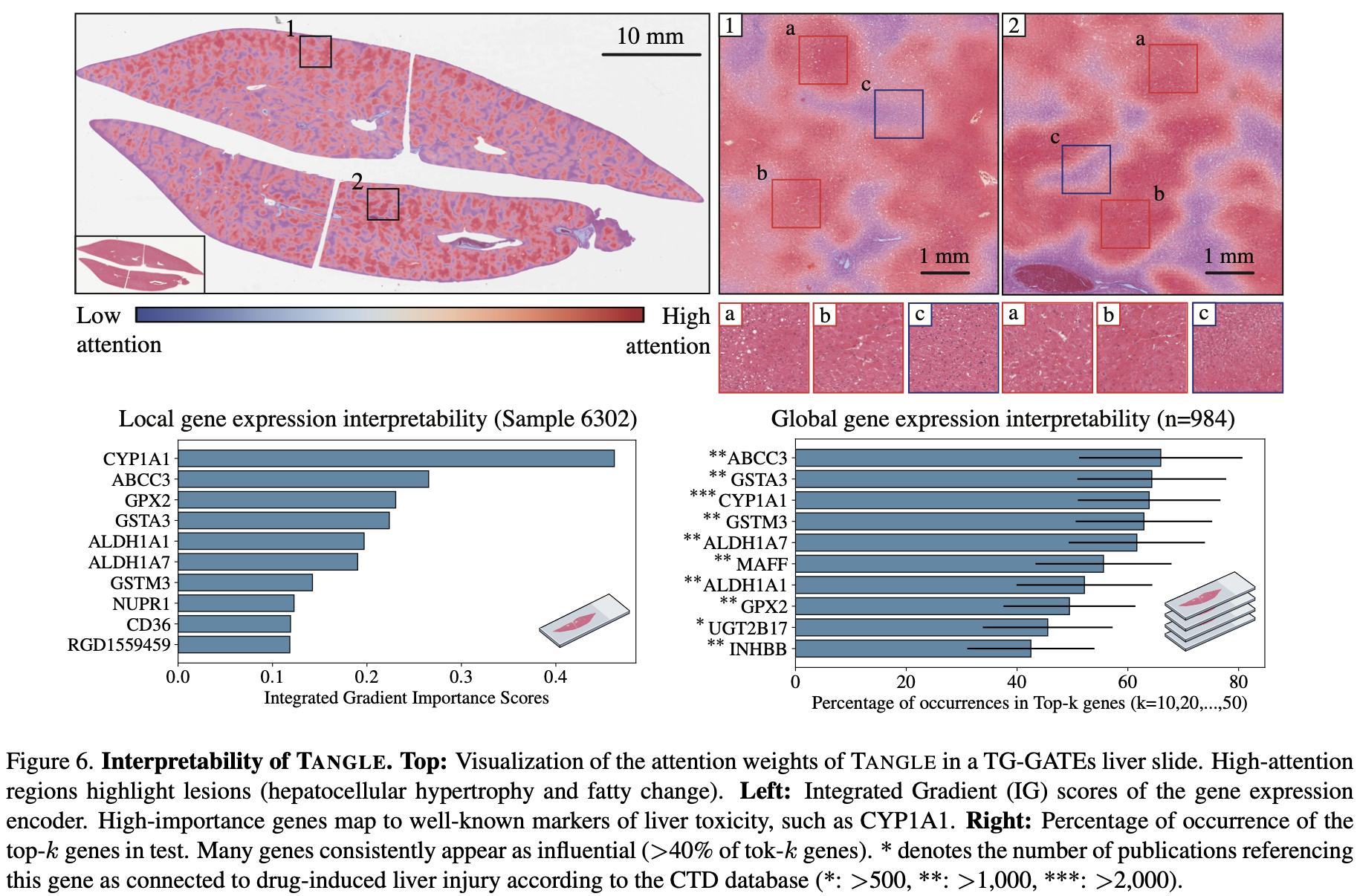
TANGLE의 interpretability에 대한 그림입니다. Attention map을 통해 TANGLE의 attention을 살펴보았고, integrated gradient important score를 통해 각 유전자에 대한 local gene expression interpretability를 확인하였습니다. 또한 global gene expression interpretability를 살펴보았습니다.
Conclusion
이렇게 SSL methodology를 활용하여 TANGLE이라는 네트워크를 구성하였고, downstream task에서 높은 성능을 보여준다는 것을 통해 representation을 잘 학습할 수 있다는 것을 확인하였습니다.
Personal opinion
- Causal interpretation of TANGLE.
- SSL은 위에서 언급하였듯 수식적으로 causal한 이론적 배경을 가지고는 있지만, 이 논문에서 이야기한 TANGLE의 성능이나 interpretability는 인과적 맥락에서 학습된 것이라거나 해석된 것이 아닙니다. 단순한 correlation을 통해 수행된 연구이기 때문에 인과적 맥락을 톺아볼 필요가 있다고 생각됩니다.
- Performance is not all.
- 본 논문은 loss의 디자인을 통한 학습이 거의 연구의 주를 이룹니다. 이는 2010년대 후반에 유행했던 연구 방법론으로, 현재는 그것보다는 더 고찰이 많이 들어간 연구 풍조가 유행해 있습니다. 본 논문도 이러한 연구 방법에 대한 고찰을 더 많이 했다면 어땠을까 하는 고민을 해봅니다.
- Slide embedding
- 그럼에도 불구하고, patch embedding을 넘어서 slide embedding space를 디자인하려는 노력은 매우 창의적입니다. 제가 병리쪽 전문가가 아니라 이러한 연구들이 이전에도 있었는지는 모르겠지만 slide-level representation을 디자인하려는 시도는 단어 그대로 brilliant하였다고 평가할 수 있습니다.
